1. 如何製備Southern blotting 用的DNA?
Lysis bffer
10 mM Tris
0.02 M EDTA
0.5% SDS
10 mg/ml RNAase
Low TE
10 mM Tris
0.1 mM EDTA
ES cell 抽取DNA
↓24 well plate 中每個 well 加入 0.5ml Lysis buffer,37℃ 水平混合 1 小時
↓準備 new eppendorf tube,每個 tube 加入 10μl Proteinase K (10mg/ml)
↓於 plate 內以 blue tip 輕輕攪拌確定 lyse 完全後 transfer 至 eppendorf tube 內 (已加 PK)
↓50℃,overnight (置於有混合裝置的培養箱反應)
↓加入 Phenol/CHCl3(1:1)0.5 ml
↓室溫,混合( on shaker),10 min
↓13200 rpm,4℃ 離心,10 min
↓準備 new eppendrof tube,每個 tube 加入 30 μl 7.5 M NH4OAC
↓將上清液移至 new eppendrof tube(已加 7.5 M NH4OAC),mix 均勻(務必確實)
↓加入 100% 酒精 1 ml,mix 均勻(有白色絲狀物出現)
↓4℃ 酒精沉澱,overnight (可省略)
↓13200 rpm,4℃,10 min
↓吸除上清液(小心!底部有白色沉澱)
↓加入 70% 酒精 1 ml wash
↓13200rpm,4℃,3~5 min
↓吸除上清液(小心!底部有白色沉澱)air dry
↓加入 Low TE ( * → 50μl 2 → 34μl 1 → 16μ)
實驗每次所需10μg (700~800 ng/μl), 請給 20μg以上
3. 是否可使用市面上賣的快速萃取DNA試劑組,來製備Southern Blot所需的DNA?
不建議,因市面上賣的試劑組(kit) 抽出來的DNA濃度較低,可能會造成Southern Blot無法順便進行。
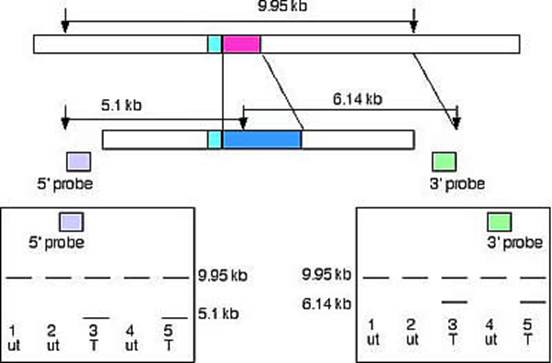
探針必需在同位互換區(homologous arm)之外,且在限制酶切點之內 (請參考圖示) ,才能辨別出 targeted 與 random integration 。
圖示:常見的三種篩選法
1.當插入的片段多出限制酶切點時

ut = untargeted T= targeted
Both probes hybridize with a 9.95 kb band at the wild type allele.
For the 5' probe, the targeting event replaces the pink exon with the blue exon containing the restriction site, creating a 5.1 kb band.
For the 3' probe, the targeting event replaces the pink exon with the blue exon containing the restriction site, creating a 6.14 kb band.
2.當插入的片段使原本的限制酶切點消失時
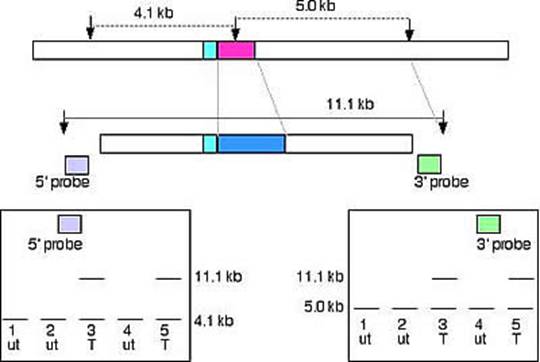
ut = untargeted
T= targeted
For the 5' probe, the wild type allele makes a 4.1 kb band. For the 3' probe, the wild type allele makes a 5.0 kb band
For both probes, the deletion of the restriction site (in the pink box) and replacement with the blue sequence lacking the site makes an 11.1 kb band.
3.當插入的片段不影響限制酶切點時
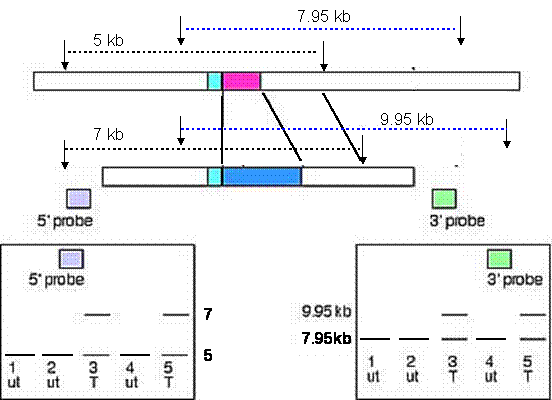
ut = untargeted T= targeted
For the 5' probe, the wild type allele makes a 5 kb band and targeted allele makes a 7 kb band. For the 3' probe, the wild type allele makes a 7.95 kb band and targeted allele makes a 9.95 kb band.
建議在300 bp~1000 bp
至少400 ng 以上(conc. >5 ng/λ),請務必將產物進行 gel purification,並附上定量圖與BLAST的結果。
請務必將PCR產物進行gel purification , 確保得到的是專一的片段。
8. 是否可只用純化管柱(Column) 來純化Probe?
只用純化管柱(Column) 所純化的Probe,在進行hybridization時容易會出現非專一性的結合,所以請務必將PCR產物進行gel purification 。
9. Southern Blot的DNA marker片段大小?
High Range DNA Ladder (kb): 48.5、24.5、20.6、17、15.3、13.8、12.1、10.2
1 Kb DNA Ladder (kb): 10、8、6、5、4、3.5、3、2.5、2、1.5、1
10. Southern Blot所使用的酵素,適合分辨的片段大小?
若限制酶酵素作用後DNA片段若是在10~17 kb之間,WT和Targeting的片段之間要大於2 kb,在17~25kb之間要大於3kb,若是10 kb以下,建議WT和Targeting的片段要大於1 kb,以利於分辨基因型,可參考第上題的DNA marker片段大小。
11. 如何選擇限制酶酵素的切點?
限制酶切點必需跨越標的載體 (targeting vector) 的同位互換區 (homologous arm)。
12. 如何確認遠端 loxp site是否有互換進入?
依據每個 case 的不同,當 User 經由 Southern 確定有篩選到正確 targeted 的細胞株後,User需在遠端 loxp site附近設計 primer,以 PCR 的方式偵測遠端 loxP site 是否存在,再以遠端 loxP 特有的限制酶 (EcoRI、BamHI、AvaI、PstI、XhoI) 切點來驗證 PCR 的產物是否正確,最後再將 PCR 產物送定序進行最後確認。
限制酶的選擇請優先參考對照表(如下表) 。
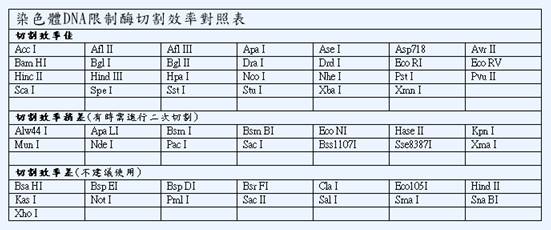
平均一個案子會篩選 24 x 8 共 192 clone。